Trong những năm gần đây, công nghệ giải trình tự gene nhằm chẩn đoán sớm và phòng chống sự lây lan bệnh dịch trong cộng đồng ngày càng thu hút các nhà khoa học. Những thành công trong xây dựng quy trình giải trình tự hệ gene virus SARS-CoV-2, quy trình phát hiện ctDNA trong giai đoạn sớm của ung thư, xác định vi khuẩn đa kháng thuốc gây nhiễm khuẩn trong bệnh viện, đã cho thấy khả năng làm chủ công nghệ của đội ngũ khoa học Việt Nam trong lĩnh vực y tế.
Giải trình tự bộ gene (hoặc giải trình tự toàn bộ bộ gene) là một xét nghiệm toàn diện, có khả năng phát hiện gần như tất cả các biến thể DNA trong bộ gene, được dùng để xác định toàn bộ cấu trúc di truyền của một sinh vật hoặc loại tế bào cụ thể. Dùng phương pháp để tìm những thay đổi trong các khu vực của bộ gene, giúp các nhà khoa học hiểu được tình trạng các bệnh cụ thể, ví dụ như ung thư. Bên cạnh đó, kết quả giải trình tự gene cũng có thể dùng để chẩn đoán và điều trị. Cho đến nay, việc giải trình tự gene cho phép chẩn đoán hơn 6.000 tình trạng bệnh và được liệt kê trong cơ sở dữ liệu Di truyền Mendel ở người (www.omim.org).
Trong chuỗi xoắn kép DNA, 4 gốc hóa học luôn có xu hướng liên kết với cùng một đối tác để tạo thành cặp bases, adenine (A) luôn bắt cặp với thymine (T) và cytosine (C) bắt cặp với guanin (G). Sự liên kết theo cặp này chính là nền tảng cho cơ chế mà các phân tử DNA được sao chép khi phân chia tế bào, đồng thời đây cũng là cơ sở cho các phương pháp thực hiện thí nghiệm giải trình tự gene.
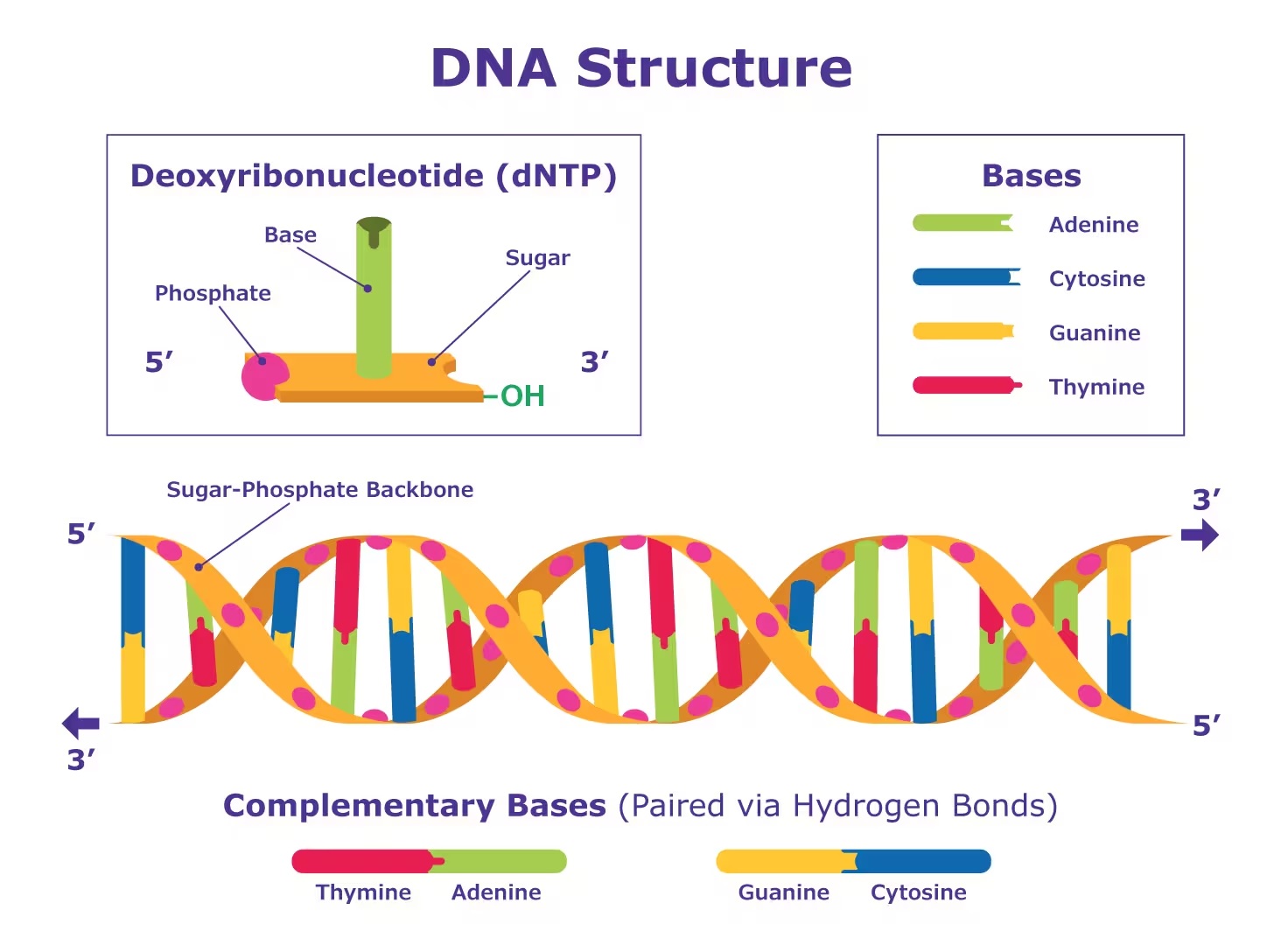
Cấu trúc DNA (Nguồn: www.sigmaaldrich.com)
Năm 1977, nhà hóa sinh học người Anh Frederick Sanger và cộng sự đã phát triển một phương pháp để giải trình tự DNA, được gọi là Phương pháp giải trình tự kết thúc chuỗi (Chain-Termination Sequencing Method - CTSM) bằng việc sử dụng các deoxy nucleotide đã bị chỉnh sửa làm mất nhóm hydroxyl ở đầu 3’ của phân tử đường. Phương pháp này dễ dàng tự động hóa, không dùng các chất độc hại nên được sử dụng phổ biến cho giải trình tự DNA từ những năm 1980 cho đến nay.
Với những tiến bộ công nghệ, trình tự bộ gene người đầu tiên đã được giải mã bằng phương pháp Sanger vào năm 2003 với sự hợp tác của 15 quốc gia do Mỹ đứng đầu, ghi nhận trình tự bộ gene người chứa khoảng 2,85 tỷ cặp bases. Kết quả hoàn thiện của nghiên cứu được công bố trong bài báo Finishing the euchromatic sequence of the human genome trên tạp chí Nature năm 2004. Tuy nhiên, nghiên cứu tiêu tốn rất nhiều thời gian và nguồn lực do phương pháp Sanger thực hiện với các đoạn DNA ngắn (khoảng 900-1.200 cặp bases), nên không hiệu quả cho các dự án có quy mô lớn. Do đó, Viện Nghiên cứu hệ gene người quốc gia (NHGRI - Mỹ) đã khởi động chương trình đầu tư với mục tiêu làm giảm chi phí giải mã hệ gene người xuống 1.000 USD trong vòng 10 năm. Đây là động lực thúc đẩy sự phát triển và thương mại hóa các công nghệ giải trình tự gene thế hệ mới.
Công nghệ giải trình tự gene thế hệ mới
Năm 2005, nền tảng công nghệ giải trình tự gene thế hệ mới (Next Generation Sequencing – NGS) đã được giới thiệu, có thể khuếch đại hàng triệu bản sao của một đoạn DNA cụ thể cùng một lúc, giúp nâng cao hiệu suất của quá trình giải mã hệ gene. Sự khác biệt chính giữa giải trình tự Sanger và NGS bắt nguồn từ khối lượng giải trình tự, NGS cho phép xử lý song song hàng triệu phản ứng, với dữ liệu đầu ra lớn, độ nhạy và tốc độ cao và tiết kiệm chi phí hơn. Rất nhiều dự án giải trình tự bộ gene mất nhiều năm với phương pháp Sanger giờ đây có thể được hoàn thành trong vòng vài giờ bằng NGS. Có thể kể đến một số công nghệ NGS đang được ứng dụng rộng rãi hiện nay như:
– Công nghệ giải trình tự Pyrosequencing 454: được phát triển bởi 454 Life Science vào năm 2005, là một hệ thống giải trình tự DNA 2 bước, có độ tương đồng cao với dung lượng lớn hơn rất nhiều so với hệ thống giải trình tự Sanger. Công nghệ pyrosequencing dựa trên nguyên lý “giải trình tự bằng tổng hợp”, bao gồm: khởi động một sợi DNA đã được giải trình tự và giải trình tự sợi bổ sung bằng phản ứng của enzyme. Hệ thống giải trình tự được 400-600 triệu cặp bases trong vòng 10 giờ, giúp giảm giá thành đáng kể so với khi sử dụng phương pháp Sanger để giải trình tự một số lượng lớn DNA.
– Công nghệ giải trình tự SBS (Sequencing By Synthesis): được phát triển bởi Solexa (sau này là Illumina) vào năm 2005, sử dụng 4 nucleotide đánh dấu huỳnh quang để giải trình tự hàng chục triệu cụm đồng thời trên bề mặt dòng chảy tế bào. Phương pháp giải trình tự SBS có khả năng xử lý đồng thời số lượng cực lớn các đoạn DNA cần đọc trình tự với độ bao phủ đồng nhất, giúp kết quả xác định biến dị di truyền có độ tin cậy cao. Khối lượng mẫu lớn cho phép sử dụng các công cụ thống kê, tương tự như các phương pháp truyền thống trong việc xác định thể đồng hợp, dị hợp và phân biệt lỗi.
– Công nghệ giải trình tự SOLiD (Sequencing by Oligonucleotide Ligation and Detection): được phát triển bởi Life Technologies và đã có mặt trên thị trường từ năm 2006. Công nghệ này được thiết kế dựa trên nguyên lý ghép nối, toàn bộ geneome được cắt thành các đoạn DNA ngắn, sau đó từng đoạn được gắn với các adapter rồi cố định vào các hạt từ. Quá trình giải trình tự được thực hiện thông qua các oligonucleotide được đánh dấu huỳnh quang. Dữ liệu của hệ thống có thể đạt khoảng hơn 1 tỷ đoạn được đọc sau mỗi lượt chạy, kích thước và độ tin cậy của mỗi đoạn DNA được đánh giá là tương đương so với hệ thống của Illumina, trong khi đó chi phí thiết bị của công nghệ SOLiD thấp hơn đáng kể so với các hệ thống khác cùng thế hệ.
– Công nghệ giải trình tự Nanoball sequencing: là một công nghệ mới, tiên tiến, được phát triển bởi Complete Genomics (sau này là Viện Genomics Bắc Kinh), có mặt trên thị trường từ năm 2009. Trong công nghệ này, các đoạn DNA được khuếch đại rồi cuộn lại thành các quả cầu nano DNA. Các nucleotide huỳnh quang liên kết với các nucleotide bổ sung và sau đó được polymer hóa để tạo ra các trình tự neo liên kết với các trình tự đã biết trên khuôn mẫu DNA. Phương pháp giải trình tự DNA này cho phép số lượng lớn các quả cầu nano DNA được giải trình tự mỗi lần chạy, với chi phí thuốc thử thấp hơn so với các nền tảng giải trình tự thế hệ tiếp theo khác.
– Công nghệ giải trình tự đơn phân tử thời gian thực (Single Molecular Real-Time): là phương pháp giải trình tự DNA phân tử đơn song song, được phát triển bởi Pacific Bioscatics, đã thương mại hóa từ năm 2011. SMRT xác định trình tự DNA bằng cách “quan sát” quá trình tổng hợp một chuỗi DNA tự nhiên bằng DNA polymerase đơn lẻ, các tín hiệu từ nucleotide được đánh dấu phosphate sẽ được phát hiện theo thời gian thực giúp xác định chính xác nucleotide nào trong 4 loại nucleotide đang được gắn vào mạch. Do đó, khi đoạn DNA được sao chép xong thì máy cũng xác định xong trình tự đoạn DNA. Công nghệ này rất hữu ích cho các ứng dụng giải trình tự toàn bộ hệ gene của các sinh vật chưa có thông tin về hệ gene.
Hiện nay, các phương pháp giải trình tự gene thế hệ mới NGS có thể giúp chẩn đoán, xác định chính xác nhiều đột biến mới của các dạng ung thư và sự xuất hiện của các dạng virus mới. Hơn nữa, các công nghệ NGS không ngừng được cải tiến, với chi phí rẻ và hiệu quả cao, ứng dụng trong chẩn đoán, có thể giúp người bệnh chủ động điều trị kịp thời.
Ứng dụng công nghệ giải trình tự gene thế hệ mới tại Việt Nam
Thời gian qua, các nhà khoa học Việt Nam đã có nhiều nghiên cứu ứng dụng công nghệ giải trình tự gene thế hệ mới trong chẩn đoán bệnh và các dịch bệnh mới xuất hiện, từ giải trình tự gene virus SARS-CoV-2 đến chẩn đoán sớm các bệnh ung thư; xác định các mối nguy hiểm từ vi khuẩn gây bệnh trong các cơ sở y tế.
Xác định nguồn gốc lây lan của virus
Nhằm đáp ứng yêu cầu xác định nguồn gốc virus trong thời gian ngắn, ứng dụng công nghệ hiện đại, nhiệm vụ KH&CN “Giải trình tự denovo virus SARS-CoV-2 gây bệnh viêm đường hô hấp cấp Covid-19 bằng hệ máy giải trình tự thế hệ mới PacBio Sequel” được Viện Công nghệ sinh học (Viện Hàn lâm Khoa học và Công nghệ Việt Nam) phối hợp với Viện Công nghệ sinh học, Viện Pasteur TP.HCM và Viện Vệ sinh dịch tễ Trung ương thực hiện từ năm 2020, và được nghiệm thu, công bố kết quả vào tháng 6/2021.
Đây là nghiên cứu đầu tiên của các nhà khoa học Việt Nam trong việc xây dựng quy trình công nghệ giải trình tự hệ gene virus SARS-CoV-2 bằng kỹ thuật giải trình tự hệ gene đoạn dài đơn phân tử theo thời gian thực (SMRT) trên hệ thiết bị giải trình tự thế hệ mới PacBio SEQUEL. Thời gian thực hiện chỉ mất khoảng 48 giờ, thay vì 72 giờ như các phương pháp khác. Quy trình được xây dựng bao gồm 6 bước: (1) Nuôi cấy và tách chiết ARN virus; (2) Tổng hợp cDNA sợi đôi từ ARN virus; (3) Chuẩn bị thư viện DNA để giải trình tự gene; (4) Giải trình tự toàn bộ hệ gene virus nCoV; (5) Lắp ráp denovo hệ gene virus; (6) Chú giải và phân tích hệ gene virus.
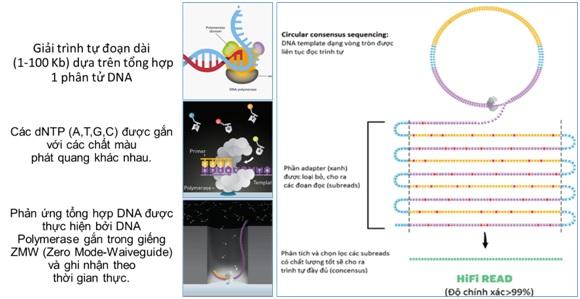
Nguyên lý công nghệ giải trình tự SMRT trên Pacbio SEQUEL (Nguồn:VAST)
Kết quả của đề tài đã giải trình tự toàn bộ hệ gene của bốn chủng virus SARS-CoV-2, với chiều dài trên 29.500 nucleotide/hệ gene. Kết quả lắp ráp hệ gene cho thấy không có lỗi đọc hay đoạn trống, kết quả giải trình tự đạt độ chính xác 99,99%.
Theo các nhà khoa học, việc xây dựng và làm chủ kỹ thuật giải trình tự hệ gene đoạn dài của PacBio đối với virus SARS-CoV-2 đã mở ra khả năng giải trình tự hệ gene virus nhanh, chính xác mà không cần dựa vào trình tự gene tham chiếu quốc tế. Trong tương lai, các nhà khoa học Việt Nam có thể ứng dụng quy trình này để giải trình tự những đối tượng virus gây bệnh mới. Việc làm chủ công nghệ cũng góp phần xác định nhanh được nguồn gốc lây lan của virus, từ đó có cơ sở khoa học trong xây dựng chiến lược, phương án phòng, chống hiệu quả sự lây lan của virus trong cộng đồng và để triển khai các phác đồ điều trị tối ưu.
Chẩn đoán ung thư đại trực tràng
Nhằm đưa ra một phương pháp phát hiện sớm ung thư đại trực tràng (UTĐTT), đề tài nghiên cứu khoa học cấp Thành phố “Xây dựng quy trình phát hiện ctDNA (circulatin tumor DNA - DNA ngoại bào mang đột biến ung thư) trong ung thư đại trực tràng giai đoạn sớm bằng công nghệ giải trình tự thế hệ mới” đã được nhóm các nhà khoa học tại Đại học Y Dược TP.HCM triển khai thực hiện từ năm 2019. Kết quả được nghiệm thu vào cuối năm 2021.
Bằng việc ứng dụng công nghệ, nhóm nghiên cứu đã tách chiết thành công cfDNA (Circulating free DNA - DNA tự do) từ mẫu huyết tương của bệnh nhân và đo được nồng độ cfDNA ở nhóm bệnh nhân UTĐTT cao hơn khoảng 1,7 lần so với nhóm người khỏe mạnh. Trong đó, xu hướng gia tăng tổng hàm lượng cfDNA phần lớn bắt nguồn từ việc gia tăng hàm lượng ctDNA trong máu, với bệnh nhân ở giai đoạn tiến triển xa của ung thư, hàm lượng ctDNA gia tăng đáng kể. Theo quy trình, mảnh DNA tự do thu nhận từ mẫu huyết tương bệnh nhân sẽ được giải trình tự trên hệ thống giải trình tự NextSeq550 để xác định sự hiện diện của ctDNA. Kết quả nghiên cứu của đề tài đã chứng minh được tính khả thi về mặt kỹ thuật của quy trình sinh thiết lỏng sàng lọc ung thư giai đoạn sớm dựa trên đột biến đặc trưng ung thư, đạt độ tin cậy tương tự như nghiên cứu trên thế giới.
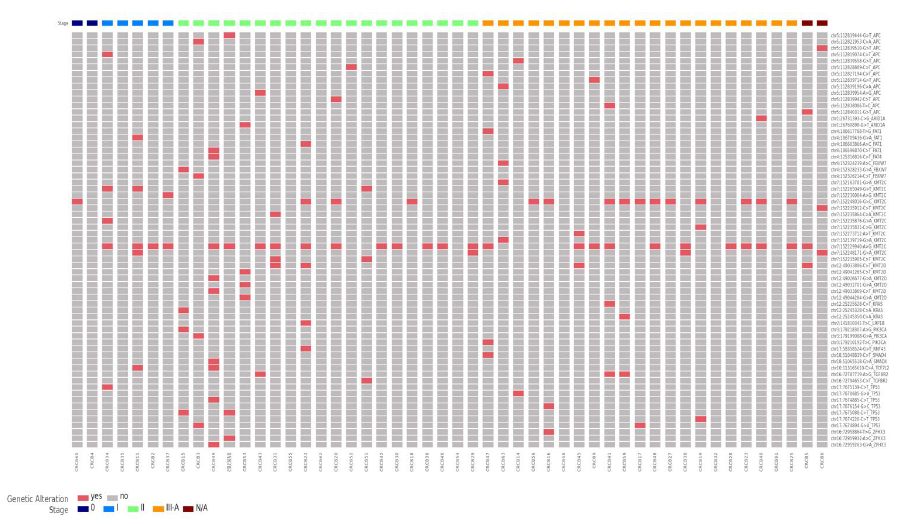
Đột biến ghi nhận trên mẫu sinh thiết lỏng của bệnh nhân UTĐTT (Nguồn: Sở KH&CN TP.HCM)
Ngoài nghiên cứu trên, Viện Di truyền Y học TP.HCM cũng đã công bố kết quả nghiên cứu “Phát hiện sớm ung thư đại trực tràng bằng công nghệ giải trình tự gene thế hệ mới” vào tháng 12/2022 trên tạp chí Future Oncology. Nghiên cứu ứng dụng quy trình sinh thiết lỏng SPOT-MAS có nền tảng dựa trên công nghệ giải trình tự gene thế hệ mới, thực hiện xét nghiệm thông qua mẫu máu để phát hiện DNA phóng thích từ tế bào khối u và lưu hành tự do trong máu của nhóm bệnh nhân UTĐTT chưa di căn, đối chiếu với DNA ngoại bào của nhóm người khỏe mạnh. Các chỉ dấu phân tử điển hình của DNA giữa hai nhóm được giải trình tự gene, phân tích và phân loại bằng mô hình học máy neuron sâu để thiết lập quy trình phân loại bệnh nhân, tiên đoán nguy cơ mắc UTĐTT.
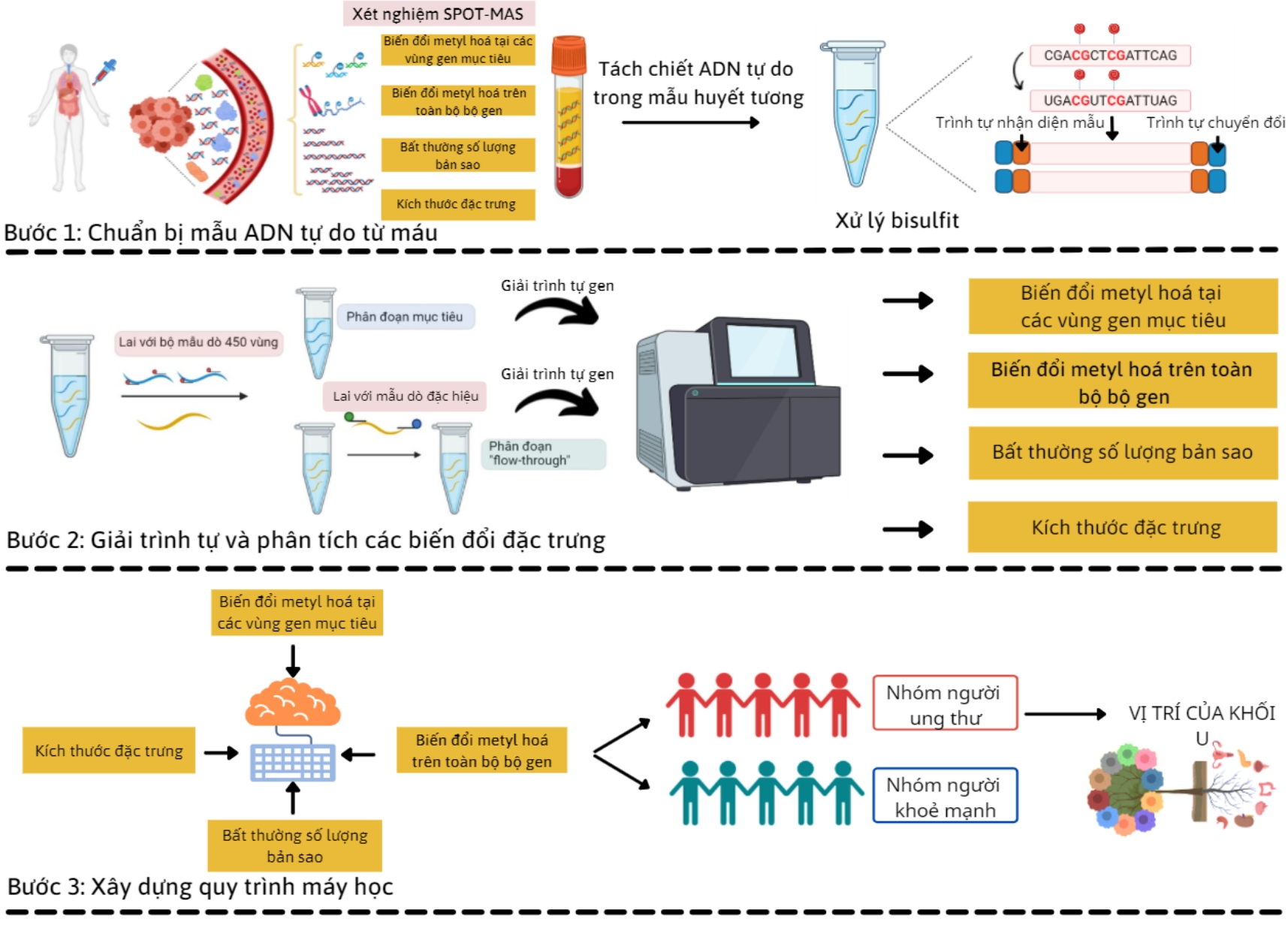
Giải pháp tầm soát công nghệ sinh thiết lỏng SPOT-MAS-ECD (Nguồn: Gene Solution)
Với độ nhạy đạt 96,8% và độ đặc hiệu 97%, nghiên cứu cho thấy SPOT-MAS có khả năng tầm soát ung thư sớm. Bên cạnh đó, nghiên cứu công bố trên tạp chí Y học Việt Nam tháng 4/2022 cũng cho thấy, phương pháp tầm soát ung thư thông qua phân tích ctDNA của công nghệ SPOT-MAS có thể sàng lọc, phát hiện sớm 5 loại ung thư phổ biến (vú, gan, phổi, dạ dày, đại trực tràng) qua một lần thu máu. Qua đó, SPOT-MAS được kỳ vọng là phương pháp xét nghiệm tầm soát ung thư không xâm lấn, là giải pháp bổ sung hiệu quả khi kết hợp với các phương pháp tầm soát ung thư thường quy.
Xác định vi khuẩn đa kháng thuốc gây nhiễm khuẩn bệnh viện
Theo ước tính của Tổ chức Y tế Thế giới vào năm 2018, với trung bình cứ 10 bệnh nhân ở Việt Nam thì có một người bị nhiễm khuẩn trong quá trình điều trị tại bệnh viện. Với thực trạng phần lớn vi khuẩn trong môi trường bệnh viện đã trở thành vi khuẩn đa kháng thuốc khó điều trị, từ năm 2016-2020, các bác sĩ tại Bệnh viện Bệnh nhiệt đới Trung ương đã phối hợp với Đại học Cambridge, Đơn vị Nghiên cứu lâm sàng Đại học Oxford, Viện Sanger Wellcome Trust (Vương quốc Anh) thực hiện đề tài KH&CN cấp Quốc gia “Ứng dụng kỹ thuật giải trình tự gene thế hệ mới để xác định vi khuẩn đa kháng thuốc (MDRO) gây nhiễm khuẩn bệnh viện”. Các nhà khoa học đã tiến hành hai nghiên cứu:
– Nghiên cứu đầu tiên về giám sát vi khuẩn kháng thuốc tại bệnh viện, thông qua việc thu thập mẫu bệnh phẩm từ bệnh nhân và môi trường bệnh viện tại các khoa hồi sức của Bệnh viện Bệnh nhiệt đới Trung ương và Bệnh viện Bạch Mai trong vòng 6 tháng. Việc giải trình tự toàn bộ hệ gene và xác định các gene kháng thuốc được thực hiện tại Viện Sanger Wellcome Trust. Kết quả cho thấy, có tới 76% bệnh nhân được lấy mẫu bị nhiễm ít nhất hai trong ba vi khuẩn đa kháng thuốc phổ biến nhất là Escherichia coli, Klebsiella pneumoniae và Acinetobacter baumannii. Kết quả này đã được công bố quốc tế trên tạp chí The Lancet Microbe vào tháng 4/2022, tại bài báo “Genomic characterisation of multidrug-resistant Escherichia coli, Klebsiella pneumoniae, and Acinetobacter baumannii in two intensive care units in Hanoi, Viet Nam: a prospective observational cohort study”.
– Nghiên cứu thứ hai thực hiện điều tra sự bùng phát của vi khuẩn đa kháng thuốc trong một ổ dịch giả định tại bệnh viện Việt Nam. Các nhà khoa học Anh đã thực hiện giải trình tự các mẫu bệnh phẩm nghi ngờ và mẫu đối chứng ngay tại Bệnh viện Bệnh nhiệt đới Trung ương. Qua đó, hỗ trợ đào tạo và giúp các bác sĩ lâm sàng và nhân viên kiểm soát nhiễm khuẩn dễ dàng ứng dụng kỹ thuật giải trình tự và tin sinh học.
Vi khuẩn đa kháng thuốc là mối đe dọa lớn đối với sức khỏe bệnh nhân (Nguồn: internet)
Kết quả nghiên cứu đặc điểm hệ gene của ba loại vi khuẩn tồn tại nhiều ở bệnh viện có thể giúp công tác vệ sinh và kiểm soát nhiễm khuẩn của bệnh viện hiệu quả hơn, có cơ sở để các bệnh viện rà soát và thay đổi các biện pháp khử khuẩn vẫn đang thực hiện. Bên cạnh đó, với sự hỗ trợ của các chuyên gia nước ngoài, các nhà khoa học tại bệnh viện đã làm chủ được kỹ thuật giải trình tự gene thế hệ mới, đủ khả năng tự thực hiện giải trình tự gene để xác định nguồn lây nhiễm vi khuẩn là từ giữa các bệnh nhân với nhau hay từ các thiết bị, dụng cụ của bệnh viện sang bệnh nhân. Từ đó, có thể đưa ra các biện pháp cách ly và diệt khuẩn phù hợp.
Với vấn nạn đáng báo động tại các bệnh viện ở Việt Nam là tình trạng vi khuẩn kháng thuốc kháng sinh, việc ứng dụng thành công công nghệ giải trình tự gene thế hệ mới giúp các nhà khoa học ở Việt Nam xác định được chính xác nguồn lây lan vi khuẩn, từ đó có thể đưa ra các biện pháp can thiệt kịp thời để cắt đứt sự lây lan, giảm tối đa nguy cơ cũng như tỷ lệ tử vong và chi phí điều trị phát sinh do vi khuẩn đa kháng thuốc gây ra cho bệnh nhân đang điều trị tại bệnh viện.
***
Có thể thấy, các nhà khoa học Việt Nam đã tiếp cận sâu với nền y học hiện đại trên thế giới, vận dụng tốt công nghệ giải trình tự gene thế hệ mới vào công tác chẩn đoán và xét nghiệm các loại virus, vi khuẩn, tế bào đột biến gây bệnh. Với nền tảng công nghệ vững chắc thể hiện qua các kết quả nghiên cứu đã được nghiệm thu, có thể tin rằng, trong tương lai sẽ có thêm nhiều nghiên cứu mới ứng dụng công nghệ giải trình tự gene, hỗ trợ ngành y chủ động hơn trong đối với các bệnh và dịch bệnh tại Việt Nam.
Duy Sang
--------------------------------------------------------------------------------
Tài liệu tham khảo chính
[1] B.Minh. Công nghệ gene giúp phát hiện DNA tế bào ung thư trong máu. https://tuoitre.vn/cong-nghe-gene-giup-phat-hien-dna-te-bao-ung-thu-trong-mau-20221025153829313.htm
[2] Biomedic. Công nghệ giải trình tự thế hệ mới - Xu hướng và ứng dụng. https://biomedic.com.vn/cong-nghe-giai-trinh-tu-the-he-moi-xu-huong-va-ung-dung/
[3] Khánh Thi. Việt Nam xây dựng thành công quy trình giải trình tự hệ gen virus SARS-CoV-2. https://dangcongsan.vn/phong-chong-dich-covid-19/viet-nam-xay-dung-thanh-cong-quy-trinh-giai-trinh-tu-he-gen-virus-sars-cov-2-582313.html
[4] Mỹ Hạnh. Xác định chính xác sự lây lan của vi khuẩn. https://khoahocphattrien.vn/khoa-hoc/xac-dinh-chinh-xac-su-lay-lan-cua-vi-khuan/20230209095246245p1c160.htm
[5] Sở KH&CN TP.HCM. Phát hiện sớm ung thư đại trực tràng bằng công nghệ giải trình tự gene thế hệ mới. https://dost.hochiminhcity.gov.vn/hoat-dong-so-khcn/phat-hien-som-ung-thu-dai-truc-trang-bang-cong-nghe-giai-trinh-tu-gene-the-he-moi/.
